概要
- パッケージとは何なのか紹介
- 具体的な使い方をコードと一緒に説明
概要
Rには操作を便利にしてくれる無数のパッケージがあります。パッケージを使うことでより簡単に高度な解析ができるようになるため今回はパッケージとはどのようなものなのか紹介したいと思います。
パッケージとは
パッケージはRの基本的な機能を補強するためのツールです。CRAN や Bioconductor などを通じてWEB上にまとめられているので、インターネットを通じて自分のパソコンに取り込んでから使います。
特定の機能を補強するものが多いため、解析作業をする中で複数のパッケージを利用することが多いです。
使い方
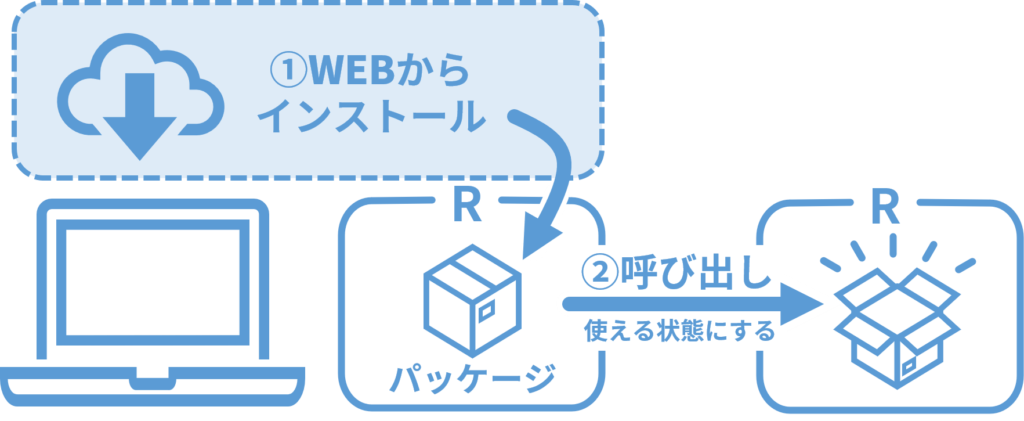
パッケージを使うためには次の2つのステップが必要です。
- パッケージのインストール
- パッケージの呼び出し
まずWebから自分のPCにパッケージを取り込み、その後取り込んだパッケージを呼び出すことで、パッケージを使うことができるようになります。
イメージは次の通りです。
具体例
パッケージを使う方法の具体例として tidyverse パッケージをインストールして使うまでの流れを紹介したいと思います。
インストール
パッケージのインストールは基本的に install.packages 関数を使います。とりあえずパッケージをインストールする時はこれを試すと良いです。
tidyverse パッケージをインストールするコードは以下のようになります。
install.packages("tidyverse")
ちなみに基本的なパッケージは install.packages 関数でインストールできますが、Bioconductor で公開されているパッケージはインストールできないことがあります。その時は BiocManager パッケージの install 関数を使ってインストールできます。
Bioconductorからパッケージをインストールする場合は以下のコードで実行できます。
# BiocManagerをインストールしてない場合は下のコードを先に実行しておく
# install.packages("BiocManager")
library(BiocManager) # BiocManagerの呼び出し
install("edgeR") # edgeR というパッケージを Bioconductor からインストール
パッケージの呼び出し
パッケージの呼び出しは library 関数で実施できます。これは CRAN からインストールしたパッケージでも Bioconductor でインストールしたパッケージでもどちらでも同じです。
tidyverse パッケージを呼び出すコードは以下のようになります。
library(tidyverse)